|
要点介绍
人类转录因子靶基因的鉴定及其数据库的构建和应用 整合大规模的人类转录因子靶基因数据,综合考虑转录因子结合位点及基因组表观修饰状态对转录因子结合的影响,采用统一预测方法预测了相对可靠的人类转录因子靶基因,并构建了开源的人类转录因子靶基因数据库,同时分析了转录因子的细胞系特异性调控、转录因子间的协作调控等。 建立了可靠的转录因子分析靶基因预测流程 分析了659个转录因子的7000多个ChIP-seq数据集,鉴定其靶基因,构建了hTFtarget人类转录因子靶基因数据库
通过对示例转录因子的靶基因预测,展示了hTFtarget的主要应用 http://bioinfo.life.hust.edu.cn/hTFtarget/
背景 转录因子(Transcription factor, TF)在细胞增殖、组织分化以及个体发育等各个阶段都起到了极其重要的作用。转录因子是基因时空表达的重要调控单元,其能结合到特定DNA基序上发挥对基因转录调控的作用。因此,检测转录因子的靶基因是研究转录因子调控的基础,而不同条件下的转录因子靶基因的变化对研究该转录因子的调控机制也有重要参考意义。染色质免疫共沉淀技术(Chromatin Immunoprecipitation,ChIP)是研究蛋白质与DNA相互作用的有力工具,而ChIP与高通量测序相结合的ChIP-seq技术,能高效地在全基因组水平上检测与组蛋白、转录因子等互作的DNA序列,在转录因子及其靶基因研究中得到广泛的应用。目前已有大量公开的转录因子ChIP-seq数据,这些数据为研究人员在全基因组水平上揭示转录因子及其靶基因在不同条件下的调控关系提供了机遇。华中科技大学生物信息与数据挖掘实验室郭安源团队整合了人类转录因子的ChIP-seq大数据,采用改进的预测方法进行靶基因预测,综合考虑和分析了表观修饰状态对转录因子结合的影响,预测了相对可靠的人类转录因子靶基因,并构建了开源的人类转录因子靶基因数据库hTFtarget(http://bioinfo.life.hust.edu.cn/hTFtarget)。 hTFtarget整合来自ENCODE、GEO和SRA数据库中659个转录因子的7190个ChIP-seq数据集、TRANSFAC、JASPAR、HOCOMOCO 的699个转录因子的高可信度DNA结合序列,采用指数衰减的BETA模型量化转录因子对靶基因的调控能力,并结合ROADMAP表观修饰状态来准确预测这些转录因子的潜在靶基因(图1A)。hTFtarget使用了ChIP-seq分析和转录因子结合位点(Transcription Factor Binding Sites, TFBS)扫描两种方法预测转录因子的靶基因。第一种方法是基于 ChIP-Seq 高通量测序的实验数据。首先,hTFtarget从ChIP-seq数据中预测该转录因子在基因组上的结合峰(Peak),并根据前1000个结合峰预测并过滤出至多 5 个可信的模体(Motif),再在所有结合峰中扫描可信模体获得转录因子的TFBS。然后根据TFBS与转录起始位点的距离,采用指数衰减的BETA模型量化转录因子对靶基因的调控能力,得到转录因子靶基因。最后,汇总每个转录因子在多个样本中的靶基因预测结果及考虑靶基因的表观修饰状态,进一步过滤得到高可信度的转录因子靶基因结果;第二种方法使用转录因子已知的结合模体预测靶基因。我们从TRANSFAC、JASPAR、HOCOMOCO 等转录因子数据库收集了699个转录因子的2737个模体的位置权重矩阵(Position Weight Matrix,PWM),通过扫描人类、小鼠和大鼠的基因组保守区域预测转录因子的潜在靶基因。同时hTFtarget还分析了转录因子的细胞系特异性调控、转录因子间的协作调控等。基于上述结果,hTFtarget提供了6个功能模块以方便用户从不同角度考察转录因子与其靶基因的调控关系(图1B)。
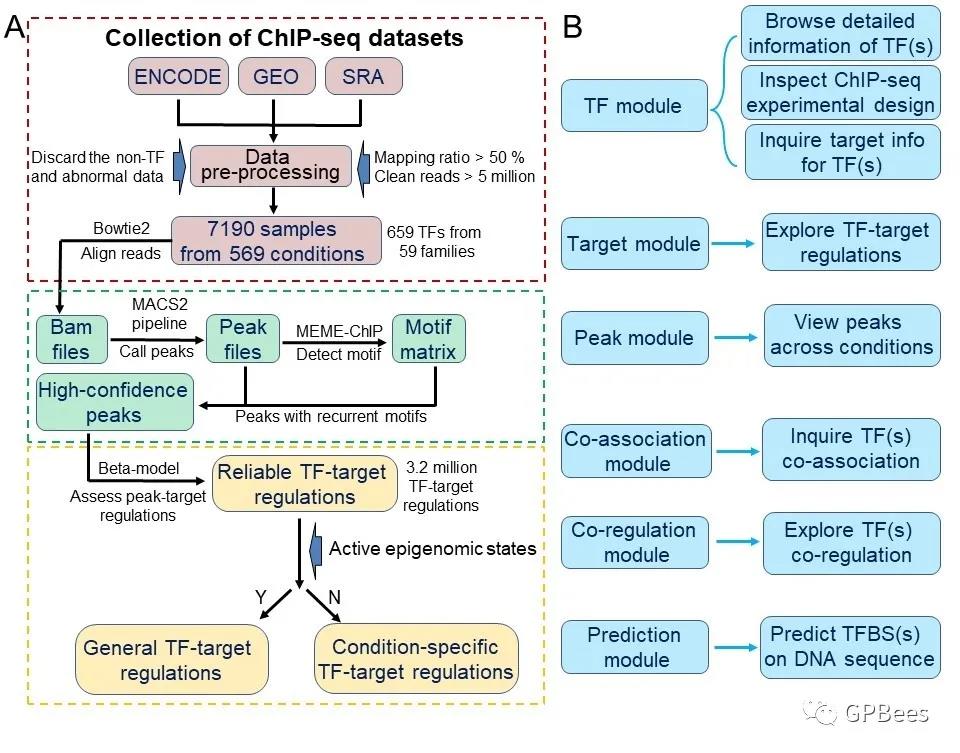
图1 hTFtarget的构建流程及主要功能模块
应用案例 1)以基因ASCL2为例,使用hTFtarget的快速检索功能浏览ASCL2作为转录因子的常态靶基因及ASCL2基因可能被哪些转录因子调控(图2A-C)。图2D-2E展示了这些调控关系(结合峰)的强弱以及该调控关系可能存在于哪些场景下(组织器官或细胞系)。 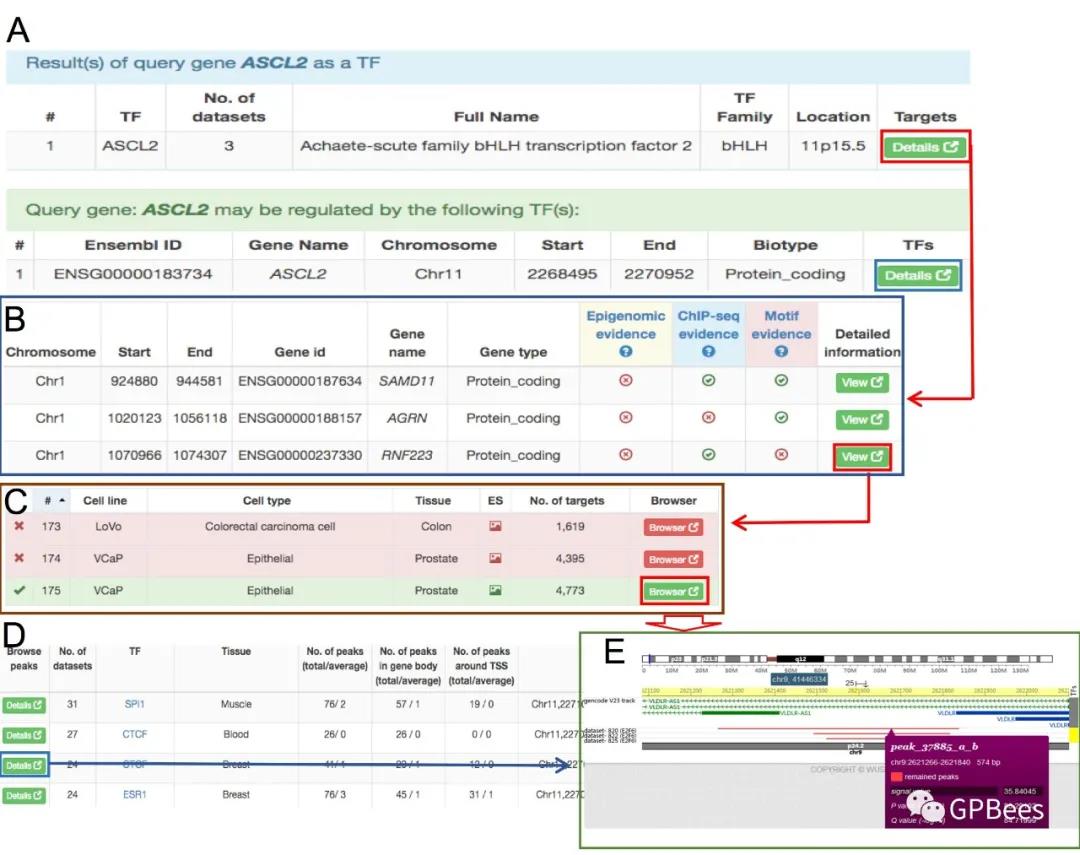
图2 hTFtarget快速检索功能一览
2)以转录因子AR为例,浏览转录因子的常态和在特定状态或组织中的靶基因(图3)。用户可以浏览hTFtarget所收集的转录因子的注释及相关ChIP-seq数据集(图3A,B)。同时可检索和查阅该转录因子所调控的特定类别基因或指定基因(图3C),并查看该调控下的结合峰强度及周边的染色质状态(图3D)。
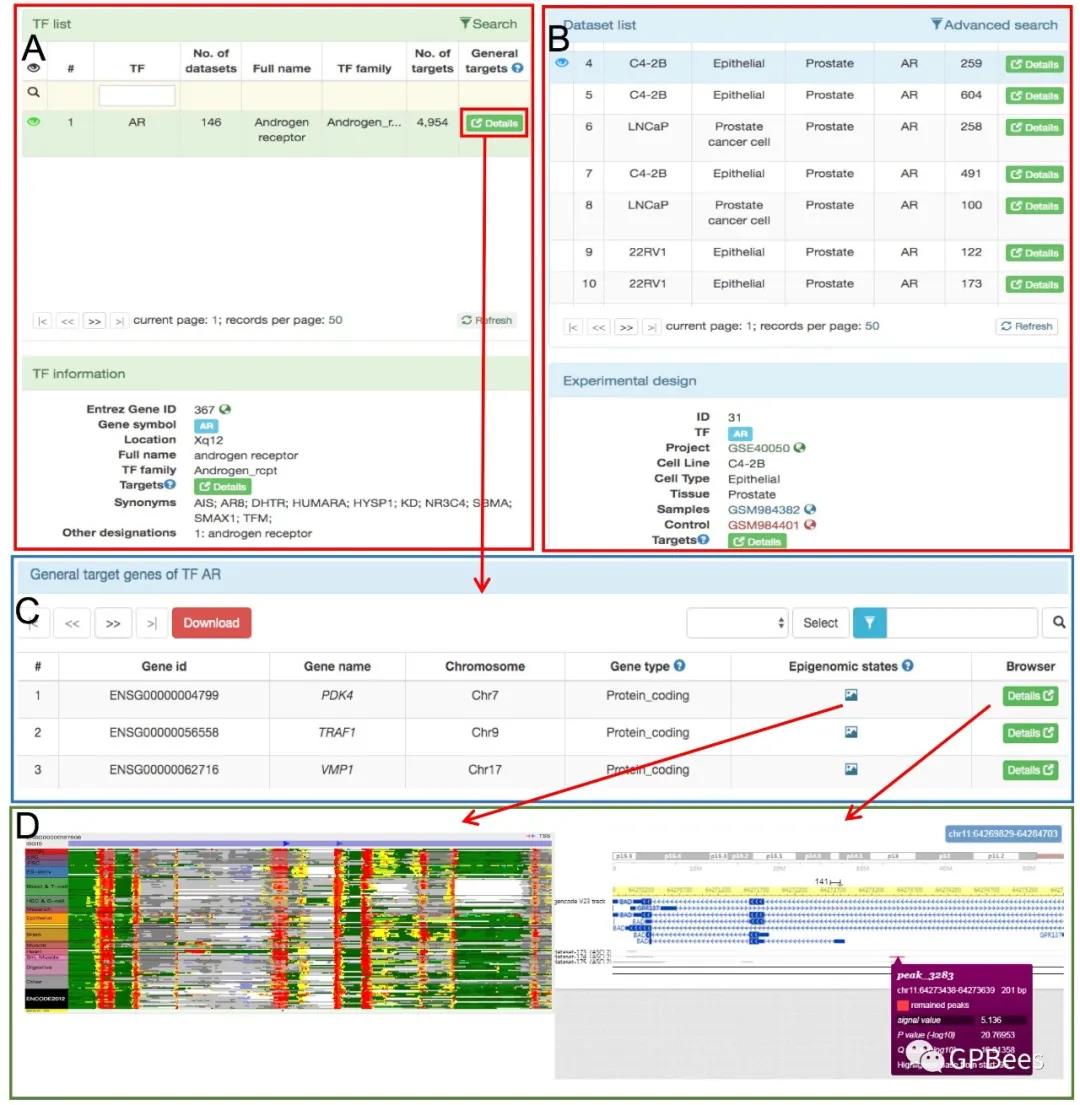
图3 hTFtarget中TF和target模块
3)hTFtarget的其他功能模块。图4A展示了hTFtarget可考察同一转录因子在不同细胞系(或同一细胞系中不同转录因子)中的结合峰状态。图4B展示了hTFtarget可检索多个转录因子对特定靶基因可能的共调控(或多个基因可被同一转录因子调控,图4B)。图4C展示了hTFtarget可预测一段DNA序列上可能的转录因子结合位点。
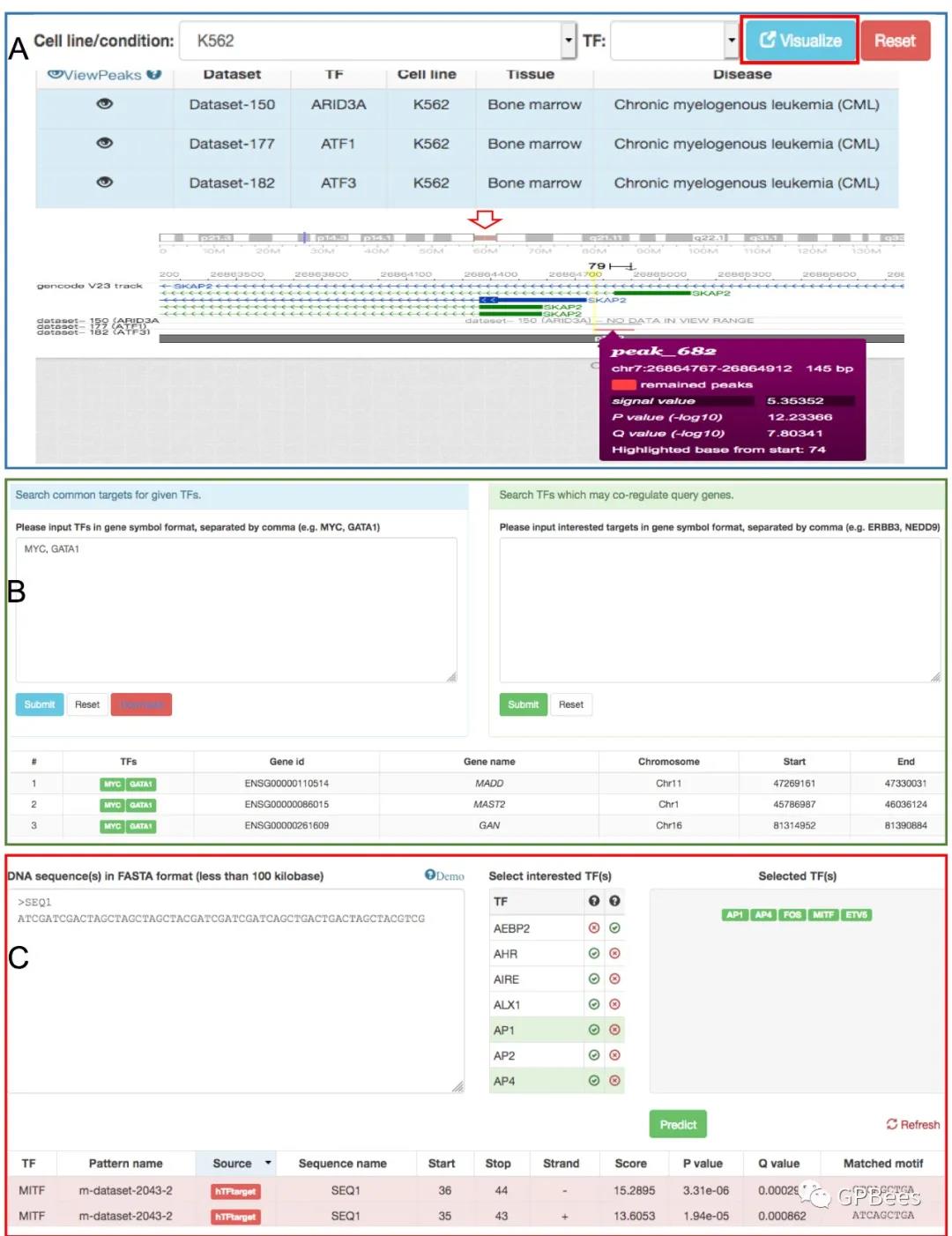
图4 hTFtarget的其他功能模块
总结和讨论
在这项研究中,郭安源团队整合了来自ENCODE、GEO和SRA数据库中转录因子的ChIP-seq数据集和现有数据库中转录因子的高可信度DNA结合序列,采用指数衰减的BETA模型量化转录因子对靶基因的调控能力,并结合ROADMAP表观修饰状态来准确预测这些转录因子的潜在靶基因(图1A),同时分析了转录因子的细胞系特异性调控和转录因子间的协作调控等。基于以上结果,该团队构建了开源的人类转录因子靶基因数据库hTFtarget,是迄今为止建立的最全的人类转录因子靶基因数据库。此外,相对于同类型其他数据库,hTFtarget还提供了全面、可信相关资源及友好的用户界面,这些资源可极大方便转录因子及基因调控相关研究领域的科研人员。hTFtarget不仅适用于单个转录因子的调控研究,还可用户发现多个转录因子之间潜在的调控网络等。hTFtarget现对学术研究人员免费开放,可通过以下链接对其收录的资源进行检索和浏览:http://bioinfo.life.hust.edu.cn/hTFtarget。
文章编译来源:Qiong Zhang, Wei Liu, Hong-Mei Zhang, Gui-Yan Xie, Ya-Ru Miao, Mengxuan Xia, et al. hTFtarget: A Comprehensive Database for Regulations of Human Transcription Factors and Their Targets. Genomics Proteomics Bioinformatics 2020;18(2).引用请参考以上格式,英文全文详见https://www.sciencedirect.com/science/article/pii/S1672022920300954。
|